Wageningen, The Netherlands
April 12, 2021

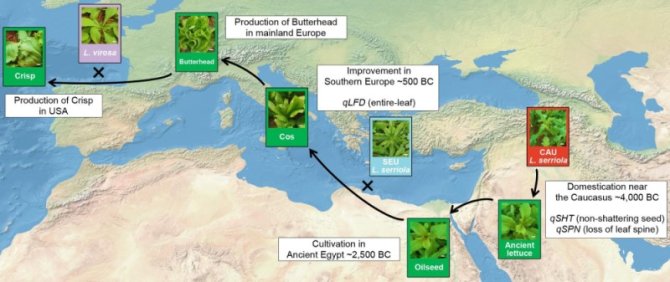
Iceberg lettuce, oakleaf lettuce, romaine, and all other lettuces that we eat nowadays, descend from wild plants that were modified 6000 years ago in the Caucasus so that plant oil could be harvested from the seeds. After the ancient Greek and Romans further bred the plants to use them as leafy vegetables, lettuce also ended up on our plates over time. The special history of lettuce has been described in detail thanks to the DNA analysis of 445 types of lettuce, conducted by Wageningen University & Research and the Chinese BGI. Their research will be published today in the authoritative periodical Nature Genetics and opens the door to faster and more effective breeding of more resilient food crops.
Try to imagine a collection of 2500 different types of lettuce: approximately 1500 varieties that were ever grown by farmers somewhere in the world and roughly 1000 populations of wild lettuce plants from roadsides and nature reserves. Then try to imagine the DNA being collected from all these types of lettuce and used to determine how the lettuce on our plate came to be. The first wild plants were modified for cultivation 6000 years ago in the Caucasus. These first lettuces were only suitable for harvesting seeds to extract oil, and the ancient Greek and Romans further bred these plants (at that time, they still had thorns on the leaves) to be used as leafy vegetables. And the story told by the DNA continues, up to the Americans that needed properties from wild varieties to change soft, smooth butter lettuce into hard, puckered iceberg lettuce. We’ve learned all that information from the DNA in these lettuce types!
 Different types of lettuce around the world
Different types of lettuce around the world
Slow Migration Through Europe
The Centre for Genetic Resources, the Netherlands (CGN), which is the Dutch gene bank and part of Wageningen University & Research (WUR), manages this collection of 2500 lettuce types. This is the largest, most complete, and best documented lettuce collection in the world.
In collaboration with the Chinese BGI, the DNA order is being determined for all 2500 types, including an analysis of genetic variants and the differences and similarities between these variants. The results from the first 445 types of lettuce have led to a publication in Nature Genetics about the origins and breeding history of the crop.
It appears that a wealth of information became available. As it turns out, the modern varieties of cultivated lettuces mostly resemble their wild predecessor Lactuca serriola from the Caucasus and the first cultivated lettuces must have been grown for seed and used for oil. The slow migration of lettuce throughout Europe via the Roman Empire, as well as the transition from seed crop to leaf crop, can also be reconstructed.
Iceberg Lettuce Versus “Ancient” Butterhead Lettuce
The study was also able to determine the point at which the more recent iceberg lettuce diverged from “ancient” butterhead lettuce in the genetic material of the wild Lactuca virosa, a fact that had long been suspected based on the genealogical data of these lettuce varieties.
Analysis of the relationship between the DNA information and traits of the cultivated lettuces shows that rigorous selection took place for traits that were desirable for production and consumption, the “domestication traits” like the absence of spines and thorns, which resulted in reduced diversity in the regions of the DNA where the genes for these traits are located. It also appears that determining the location of several genes in the DNA is possible by analysing the relationship between DNA variation and traits through so-called Genome Wide Association Studies (GWAS).
The Key to a Wealth of Genetic Material for Breeding
According to Rob van Treuren and Theo van Hintum, the two Wageningen co-authors of the publication, the research beautifully demonstrates how much information can be collected from DNA information in a genebank collection. It also shows how important the preservation and protection of biodiversity and genetic sources are for a sustainable food supply in times of climate change and a growing global population.
“Determining the DNA order of the material, in our collections and others, enables science to trace the traits hidden until now, in thousands of varieties and wild populations of lettuce and other crops. In doing so, we have obtained the key to an enormous treasure chest. For instance, imagine that research indicates that certain genes are important for resistance against drought or a certain disease. Then you would be able to search in the DNA data for genetic resources that have genes that look very similar and, using those resources, you could breed plants much quicker and more effective than what was previously possible. That is nothing short of revolutionary.”
Read the publication:
- Tong Wei, Treuren, R van, …, Huan Liu (2021) Whole-genome resequencing of 445 Lactuca accessions reveals the domestication history of cultivated lettuce. Nature Genetics, doi:10.1038/s41588-021-00831-0.
Het DNA van sla ontrafeld: in 6000 jaar van onkruid tot geliefde groente
IJsbergsla, eikenblad-, Romaine en alle andere sla die wij tegenwoordig eten stamt af van wilde plantjes die 6000 jaar geleden in de Kaukasus werden aangepast zodat uit het zaad van de plant olie kon worden gewonnen. Nadat de oude Grieken en Romeinen de plantjes verder veredelden om ze als bladgroente te gebruiken, belandde sla in de loop der tijd ook op óns bord. De bijzondere ontstaansgeschiedenis van sla is gedetailleerd in kaart gebracht dankzij een DNA-analyse van 445 slasoorten, uitgevoerd door Wageningen University & Research en het Chinese BGI. Hun onderzoek wordt vandaag gepubliceerd in het gezaghebbende tijdschrift Nature Genetics en opent de deur naar snellere en effectievere veredeling van weerbaardere voedingsgewassen.
Probeer je eens voor te stellen, een verzameling van 2500 verschillende soorten sla: ongeveer 1500 rassen die ooit ergens ter wereld door boeren verbouwd werden, en nog eens ongeveer 1000 populaties wilde sla planten uit bermen en natuurgebieden. En probeer je dan voor te stellen dat uit al die slasoorten DNA wordt gehaald en dat aan de hand van dat DNA kan worden bepaald hoe de sla op ons bord is ontstaan. Dat de eerste wilde plantjes 6000 jaar geleden in de Kaukasus werden aangepast om verbouwd te worden. Dat deze eerste sla alleen geschikt was voor het oogsten van zaad waaruit olie werd gewonnen, en dat de oude Grieken en Romeinen deze plantjes (toen nog met doorns op de bladeren) verder veredelden om als bladgroente gebruikt te worden. En het verhaal dat het DNA vertelt gaat door, tot aan de Amerikanen die eigenschappen uit wilde soorten nodig hadden om van de zachte gladde botersla harde gebobbelde ijsbergsla te maken. En dat allemaal uit informatie van het DNA van deze slasoorten!
Langzame migratie door Europa
Het Centrum voor Genetische Bronnen, Nederland (CGN), dit is de Nederlandse genenbank en deel van Wageningen University & Research (WUR), beheert die collectie van 2500 slasoorten. Dit is de grootste, meest complete en best gedocumenteerde slacollectie ter wereld. In samenwerking met het Chinese BGI wordt van alle 2500 soorten de DNA-volgorde bepaald, inclusief een analyse van genetische varianten en de verschillen en overeenkomsten tussen deze varianten. De resultaten van de eerste 445 slasoorten hebben nu geleid tot een publicatie in Nature Genetics over de ontstaans- en veredelingsgeschiedenis van het gewas.
Er lijkt een schatkamer aan informatie te zijn geopend. Zo blijkt dat de diversiteit van de huidige gecultiveerde sla het meest lijkt op die van de wilde voorloper Lactuca serriola uit de Kaukasus, en dat de eerste gecultiveerde sla oliesla moet zijn geweest, geteeld voor het zaad. Ook de langzame migratie van sla, via het Romeinse rijk, door Europa kan worden gereconstrueerd, evenals de overgang van zaadgewas naar bladgewas.
IJsbergsla versus ‘ouderwetse’ botersla
De studie blijkt ook in staat te zijn het verschil tussen de recente ijsbergsla en de ‘ouderwetse’ botersla terug te brengen tot genetisch materiaal dat afkomstig blijkt van de wilde Lactuca virosa, een feit dat al vermoed werd op basis van afstammingsgegevens van deze slasoorten.
Analyse van de samenhang tussen de DNA gegevens en eigenschappen van de gecultiveerde sla leert dat er sterk is geselecteerd op eigenschappen die aantrekkelijk waren voor productie en consumptie, de zogenaamde domesticatie-eigenschappen, zoals het ontbreken van stekels en doornen, wat resulteerde in een vernauwing van de diversiteit in de regio’s in het DNA waar de genen voor deze eigenschappen voorkomen. Ook blijkt het mogelijk de positie van een aantal eigenschappen in het DNA te bepalen door de samenhang tussen DNA-varianten en deze eigenschappen te analyseren via zogenaamde Genome Wide Association Studies (GWAS).
Sleutel tot schatkist aan genetisch materiaal voor veredeling
De studie toont volgens Rob van Treuren en Theo van Hintum, de twee Wageningse co-auteurs van de publicatie, prachtig aan hoeveel informatie uit DNA-gegevens van een genenbankcollectie kan worden gehaald. Ook laat het zien hoe belangrijk het behoud en beschermen van biodiversiteit en genetische bronnen is voor een duurzame voedselvoorziening in tijden van klimaatverandering en een groeiende wereldbevolking.
“Het bepalen van de DNA-volgorde van het materiaal in onze en andere collecties stelt de wetenschap in staat om de tot nu toe verborgen eigenschappen in duizenden rassen en wilde populaties van sla en andere gewassen op te sporen. We hebben daarmee de sleutel tot een enorme schatkamer in handen gekregen. Stel je bijvoorbeeld voor dat onderzoek laat zien dat bepaalde genen belangrijk zijn voor weerstand tegen droogte of een bepaalde ziekte. Dan kun je vervolgens in de DNA-data op zoek naar gewassoorten die genen hebben die er sprekend op lijken en met die soorten veel sneller en effectiever dan tot nu toe mogelijk is gaan veredelen. Dat is niets minder dan een revolutie.”