Paris, France
July 28, 2008
Sources:
INRA -
CEA/Genoscope -
CNRS
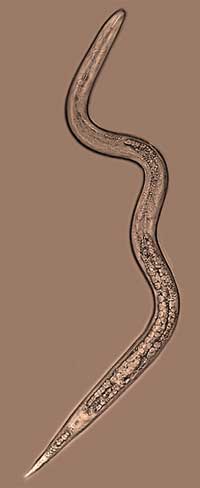 Le
premier génome d’un animal parasite de plantes vient d’être
séquencé par un consortium international de 27 laboratoires,
impliquant l’INRA, le CNRS et le Génoscope et coordonné par une
équipe de chercheurs du Centre INRA de Sophia-Antipolis. Il
s’agit du génome d’un ver parasite de plantes, le nématode à
galles, Meloidogyne incognita. Cette avancée permet de
lever le voile sur l’arsenal et les adaptations développés par
les ravageurs des cultures pour attaquer les plantes. Cette
étude a aussi conduit à l’identification de nouvelles cibles
pour le développement de stratégies de lutte spécifique contre
ces organismes. Le détail de ces travaux est publié dans
l’édition avancée en ligne de la revue « Nature Biotechnology »
du 27 juillet 2008. Le
premier génome d’un animal parasite de plantes vient d’être
séquencé par un consortium international de 27 laboratoires,
impliquant l’INRA, le CNRS et le Génoscope et coordonné par une
équipe de chercheurs du Centre INRA de Sophia-Antipolis. Il
s’agit du génome d’un ver parasite de plantes, le nématode à
galles, Meloidogyne incognita. Cette avancée permet de
lever le voile sur l’arsenal et les adaptations développés par
les ravageurs des cultures pour attaquer les plantes. Cette
étude a aussi conduit à l’identification de nouvelles cibles
pour le développement de stratégies de lutte spécifique contre
ces organismes. Le détail de ces travaux est publié dans
l’édition avancée en ligne de la revue « Nature Biotechnology »
du 27 juillet 2008.
Les nématodes sont des vers qui représentent une large fraction
de la diversité biologique sur terre, et qui colonisent tous les
milieux : 25 000 espèces sont décrites, et on estime que le
nombre total d’espèces pourrait atteindre plus d’un million. En
dehors de l’espèce Caenorhabditis elegans, qui est un modèle
pour les recherches sur le développement et le vieillissement
des organismes, très peu de choses sont actuellement connues sur
la diversité au sein du groupe des nématodes.
Les nématodes parasites de plantes, rencontrés majoritairement
dans le sol, sont responsables de dégâts estimés à plusieurs
dizaines de milliards d’euros par an à travers le monde. La
lutte chimique représentait jusqu’à une date très récente le
moyen de lutte le plus largement répandu. En raison de leur
toxicité pour la santé humaine et l’environnement, la plupart
des molécules utilisées ont été interdites récemment et la mise
au point de solutions alternatives est un enjeu majeur à très
court terme.
La publication de la séquence du génome du nématode à galles*,
Meloidogyne incognita, représente une avancée majeure à plus
d’un titre. Il s’agit en effet, à la fois, du premier séquençage
de génome d’un animal parasite de plantes et d’un métazoaire
parthénogénétique (c’est-à-dire se reproduisant sans sexualité).
Pour Pierre Abad, directeur de l’unité mixte de recherche
Interactions biotiques en santé végétale (INRA-CNRS-Université
de Nice Sophia-Antipolis) et coordonnateur de ce projet, « cette
étude va permettre d’avoir un premier éclairage sur l’arsenal
parasitaire présent chez ces ravageurs de plantes et sur les
mécanismes génétiques à l’origine de la formidable capacité
d’adaptation de ces organismes asexués. »
Le Génoscope (Paris, France) a produit au total plus de 1
million de fragments séquencés à partir desquels la prédiction
de gènes a été réalisée avec le support de l’INRA de Toulouse.
Les ressources et les compétences de l’ensemble des partenaires
du consortium international (dont l’INRA, le CNRS, et le
Génoscope, pour la France) ont ensuite été mobilisées pour
analyser l’ensemble de ces données.
Un génome complexe à l’origine d’une extrême plasticité
Au cours de l’analyse du génome, les chercheurs ont eu la
surprise de constater que le génome de ce nématode était en fait
constitué de la juxtaposition d’au moins deux génomes. Le taux
de divergence moyen entre ces différents génomes est un des plus
importants jamais observé jusqu’à présent dans le règne vivant.
Cette particularité génétique pourrait être à l’origine de
l’acquisition rapide de nouvelles fonctions conférant à ces
organismes asexués leur grande capacité d’adaptation responsable
de leur large distribution à travers la planète.
Un arsenal enzymatique adapté au parasitisme des plantes
Un autre résultat majeur de cette analyse est l’identification
d’un large éventail enzymatique permettant la dégradation de la
paroi cellulaire des plantes. Cette diversité et cette richesse
sont d’une extrême originalité dans la mesure où elles restent
inégalées à ce jour dans le règne animal. La nature des gènes
impliqués est également surprenante puisqu’ils présentent de
très fortes similitudes avec des gènes bactériens, suggérant
l’existence de multiples évènements de transferts horizontaux
d’origine bactérienne. L’acquisition de nouvelles fonctions via
les transferts de gènes pourrait représenter un élément clé de
l’adaptation des organismes animaux au parasitisme des plantes.
L’analyse comparée du génome de ce nématode avec ceux d’autres
nématodes, comme C. elegans et le nématode parasite de l’homme
Brugia Malayi ou encore la drosophile, a également permis
d’avoir une première image de ce qui fait l’identité des
parasites animaux, mettant en évidence par là-même de nouvelles
cibles potentielles, plus spécifiques, pour lutter contre ces
organismes.
Cette première séquence génomique complète d’un organisme animal
parasite de plantes apporte donc des bases pour la compréhension
des relations hôte-pathogène et elle complète notre vision des
adaptations par lesquelles les ravageurs de plantes envahissent
leurs hôtes.
Dans un avenir proche, le séquençage programmé d’autres génomes
de nématodes ayant des modes de vie différents permettra de
mieux comprendre le succès évolutif de ces organismes à
l’échelle de la planète.
* Meloidogyne incognita est un parasite extrêmement
polyphage, capable d’attaquer plus de 3000 plantes hôtes. Il est
particulièrement dommageable sur les cultures maraîchères
(tomate, piment, melon, etc.), sur le café, le coton, etc.
Référence
Genome sequence of the
metazoan plant-parasitic nematode Meloidogyne incognita
Nature
Biotechnology
Advance Online Publication 27 juillet 2008, DOI :
10.1038/nbt.1482
http://www.nature.com/nbt/index.html
Pierre Abad1,2,3*, Jérôme Gouzy4, Jean-Marc Aury5,6,7,
Philippe Castagnone-Sereno1,2,3, Etienne G.J. Danchin1,2,3,
Emeline Deleury1,2,3, Laetitia Perfus-Barbeoch1,2,3,
Véronique Anthouard5,6,7, François Artiguenave5,6,7, Vivian
C. Blok8, Marie-Cécile Caillaud1,2,3, Pedro M. Coutinho9,
Corinne Dasilva5,6,7, Francesca De Luca10, Florence
Deau1,2,3, Magali Esquibet11, Timothé Flutre12, Jared V.
Goldstone13, Noureddine Hamamouch14, Tarek Hewezi15, Olivier
Jaillon5,6,7, Claire Jubin5,6,7, Paola Leonetti10, Marc
Magliano1,2,3, Tom R. Maier15, Gabriel V. Markov16,17, Paul
McVeigh18, Graziano Pesole19,20, Julie Poulain5,6,7, Marc
Robinson-Rechavi21,22, Erika Sallet23,24, Béatrice
Ségurens5,6,7, Delphine Steinbach12, Tom Tytgat25, Edgardo
Ugarte5,6,7, Cyril van Ghelder 1,2,3, Pasqua Veronico10,
Thomas J. Baum15, Mark Blaxter26, Teresa Bleve-Zacheo10,
Eric L. Davis14, Jonathan J. Ewbank27, Bruno Favery1,2,3,
Eric Grenier11, Bernard Henrissat9, John T. Jones8, Vincent
Laudet16, Aaron G. Maule18, Hadi Quesneville12, Marie-Noëlle
Rosso1,2,3, Thomas Schiex24, Geert Smant25, Jean
Weissenbach5,6,7, Patrick Wincker5,6,7
1INRA, UMR 1301, F-06903 Sophia-Antipolis, France. 2CNRS,
UMR 6243, F-06903 Sophia-Antipolis, France. 3UNSA, UMR 1301,
F-06903 Sophia-Antipolis, France. 4Laboratoire Interactions
Plantes Micro-organismes UMR441/2594, INRA/CNRS, F-31320
Castanet Tolosan. 5Genoscope (CEA), 2 rue Gaston Crémieux
CP5706, 91057 Evry, France. 6CNRS, UMR 8030, 2 rue Gaston
Crémieux CP5706, 91057 Evry, France. 7Université d'Evry,
91057 Evry, France. 8Plant Pathology Programme, SCRI,
Invergowrie, Dundee, DD2 5DA, UK. 9CNRS, UMR 6098 CNRS and
Universités d'Aix-Marseille I & II, Marseille, France.
10Istituto per la Protezione delle Piante, Consiglio
Nazionale delle Ricerche, Via G. Amendola 165/a, 70126,
Bari, Italy. 11INRA, Agrocampus Rennes, Univ Rennes I,
UMR1099 BiO3P; F-35653 Le Rheu;France. 12INRA, UR1164 Unité
de Recherche en Génomique et Informatique (URGI), 523 place
des terrasses de l'Agora 91034 Evry, France. 13Biology
Department, Woods Hole Oceanographic Institution, Woods
Hole, MA, USA. 14Department of Plant Pathology North
Carolina State University 840 Method Road, Unit 4, Box 7903
Raleigh, NC 27607. 15Department of Plant Pathology, Iowa
State University, 351 Bessey Hall, Ames, IA 50011, USA.
16Université de Lyon, Institut de Génomique Fonctionnelle de
Lyon, Molecular Zoology team, Ecole Normale Supérieure de
Lyon, Université Lyon 1, CNRS, INRA, Institut Fédératif 128
Biosciences Gerland Lyon Sud, France. 17USM 501 - Evolution
des Régulations Endocriniennes. Muséum National d'Histoire
Naturelle, Paris, France. 18Biomolecular Processes:
Parasitology, School of Biological Sciences, Medical Biology
Centre, 97 Lisburn Road, Queen's University Belfast,
Belfast, BT9 7BL, UK. 19Dipartimento di Biochimica e
Biologia Molecolare "E. Quagliariello", University of Bari,
Italy. 20Istituto Tecnologie Biomediche, Consiglio Nazionale
delle Ricerche, Bari, Italy. 21Department of Ecology and
Evolution, University of Lausanne, Switzerland. 22Swiss
Institute of Bioinformatics, 1015 Lausanne, Switzerland.
23Plateforme Bioinformatique du Génopole Toulouse
Midi-Pyrénées, GIS Toulouse Genopole, F-31320 Castanet
Tolosan. 24Unité de Biométrie et d'Intelligence Artificielle
UR875, INRA, F-31320 Castanet Tolosan. 25Laboratory of
Nematology, Wageningen University, Binnenhaven 5, 6709PD
Wageningen, The Netherlands. 26Institute of Evolutionary
Biology, University of Edinburgh, Edinburgh EH9 3JT, UK.
27INSERM/CNRS/Université de la Méditerranée, Centre
d'Immunologie de Marseille-Luminy, F-13288, France.
Genome sequence
of the metazoan
plant-parasitic
nematode
Meloidogyne
incognita
Pierre Abad, Jérôme
Gouzy, Jean-Marc
Aury, Philippe
Castagnone-Sereno,
Etienne G J Danchin,
Emeline Deleury,
Laetitia
Perfus-Barbeoch,
Véronique Anthouard,
François
Artiguenave, Vivian
C Blok, Marie-Cécile
Caillaud, Pedro M
Coutinho, Corinne
Dasilva, Francesca
De Luca, Florence
Deau, Magali
Esquibet, Timothé
Flutre, Jared V
Goldstone,
Noureddine
Hamamouch, Tarek
Hewezi, Olivier
Jaillon, Claire
Jubin, Paola
Leonetti, Marc
Magliano, Tom R
Maier, Gabriel V
Markov, Paul
McVeigh, Graziano
Pesole, Julie
Poulain, Marc
Robinson-Rechavi,
Erika Sallet,
Béatrice Ségurens,
Delphine Steinbach,
Tom Tytgat, Edgardo
Ugarte, Cyril van
Ghelder, Pasqua
Veronico, Thomas J
Baum, Mark Blaxter,
Teresa Bleve-Zacheo,
Eric L Davis,
Jonathan J Ewbank,
Bruno Favery, Eric
Grenier, Bernard
Henrissat, John T
Jones, Vincent
Laudet, Aaron G
Maule, Hadi
Quesneville,
Marie-Noëlle Rosso,
Thomas Schiex, Geert
Smant, Jean
Weissenbach &
Patrick Wincker
Published online:
27 July 2008;
|
doi:10.1038/nbt.1482
Abstract
:
www.nature.com/nbt/journal/vaop/ncurrent/abs/nbt.1482.html |
|
|